DYNAMICS™ Vorteile
Größe und Größenverteilungen
Molare Masse
Zeta Potential
Partikel-Konzentration
Thermische Stabilität
Kolloidale Stabilität
Konzentrationsanalyse
Parametrische Analyse
Viskositätsberechnungen
Trübung oder Opaleszenz
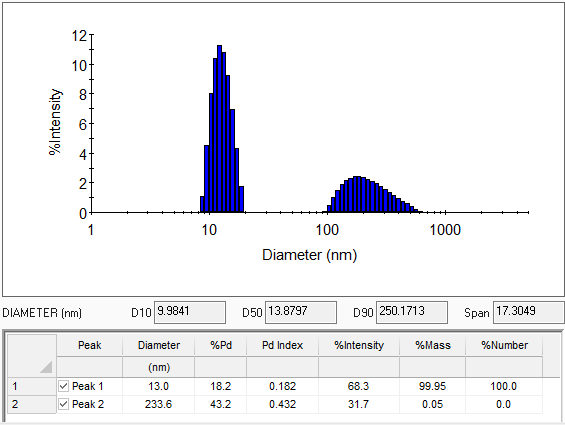
Größe und Größenverteilungen - die Grundlagen
Bestimmen Sie den durchschnittlichen Radius bzw. Durchmesser oder Diffusionskoeffizienten und deren Verteilung. Die Verteilung kann als Polydispersität, prozentuale Polydispersität oder Polydispersitätsindex (PDI), alle Parameter gemessen und angezeigt werden.
Die Kumulantenmethode bestimmt die durchschnittliche Größe und Polydispersität. DYNAMICS ermittelt die Partikelgröße gemäß dem in ISO 22412 und ASTM490-09 spezifizierten Kumulantenalgorithmus. Alternativ können Sie eine Wyatt-eigene Kumulantenberechnung für eine weiterentwickelte Auswertung wählen, die realitätsnähere Ergebnisse liefert.
Eine Probe mit hoher Polydispersität weist auf das Vorhandensein von Aggregaten oder mehreren Spezies hin. Die Regularisierung, ein Algorithmus der nicht-negativen kleinsten Quadrate (NNLS), löst Populationen auf, die sich um den Faktor 3-5 in der Größe unterscheiden. Verteilungen können als %Intensitäts-, %Massen- oder %Anzahl-Verteilung berechnet und angezeigt werden.
Bei einer breiten Größenverteilung ist es wichtig, die gesamte Verteilung zu verstehen und nicht nur den mittleren Radius. DYNAMICS bietet die Berechnung der kumulativen Größenverteilung (entweder Durchmesser oder Radius), mit Werten für D10, D50 und D90 (die Größe der Probe kleiner als 10 %, 50 % bzw. 90 % der gesamten Verteilung). Die Breite der Verteilung, definiert als (D90-D10)/D50, wird ebenfalls berechnet.
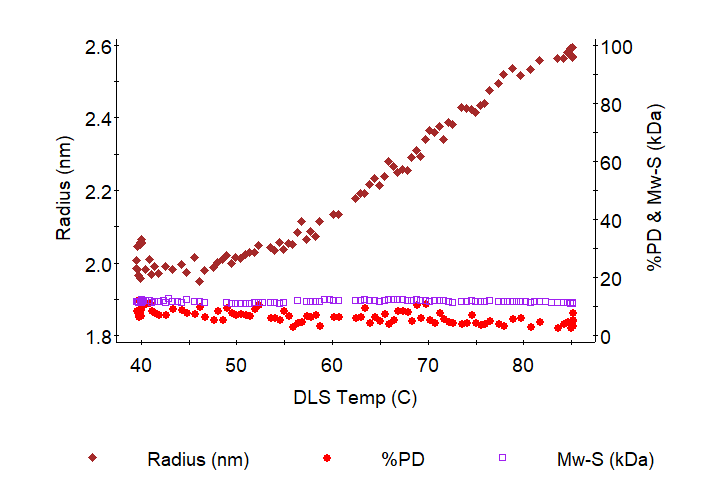
Molare Masse durch statische Lichtstreuung
Messen Sie die absolute molare Masse (Molekulargewicht) von Proteinen und kleinen Polymeren in Ihrem DynaPro™ Plate Reader III oder DynaPro™ NanoStar™, gleichzeitig mit DLS.
Die Kombination aus Molmassen- und Größenmessungen, die gleichzeitig durchgeführt werden, bestimmt die molekulare Konformation und kann zwischen Größenänderungen aufgrund von Proteinentfaltung und -aggregation unterscheiden.
Die Grafik zeigt ein Beispiel für die Entfaltung von Proteinen. Der mittels DLS gemessene Radius ändert sich von 2 auf 2,6 nm. Die konstante molare Masse und Polydispersität (%PD) von 40 bis 85 °C deuten darauf hin, dass die Größenzunahme bei Erwärmung auf Entfaltung und nicht auf Aggregation zurückzuführen ist.
Für Messungen der absoluten Molmassenverteilung oder hochmolekularer Polymere empfehlen wir den Einsatz unserer Mehrwinkel-Lichtstreuungsgeräte gekoppelt mit Größenausschlusschromatographie, SEC-MALS.
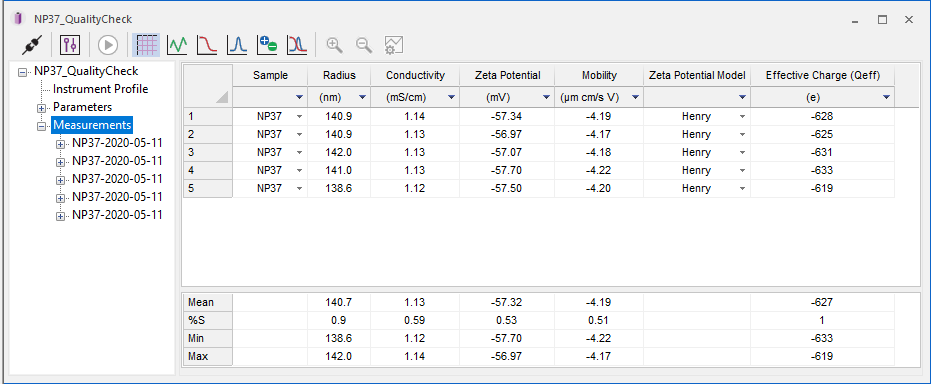
Zeta-Potential und Ladung
Bei der elektrophoretischen Lichtstreuung muss DYNAMICS keine Kompromisse eingehen. Es unterstützt die gängigsten Modelle Henry, Hückel und Smoluchowski zur Berechnung des Zetapotentials in Abhängigkeit von Proben- und Lösungsbedingungen.
In Kombination mit Rh aus DLS (gleichzeitig gemessen) werden auch die Netto- und effektive Ladung auf einem Partikel berechnet.
Darüber hinaus bestimmt DYNAMICS die Leitfähigkeit, die Debye-Länge und den Wert der Henry'schen Funktion.
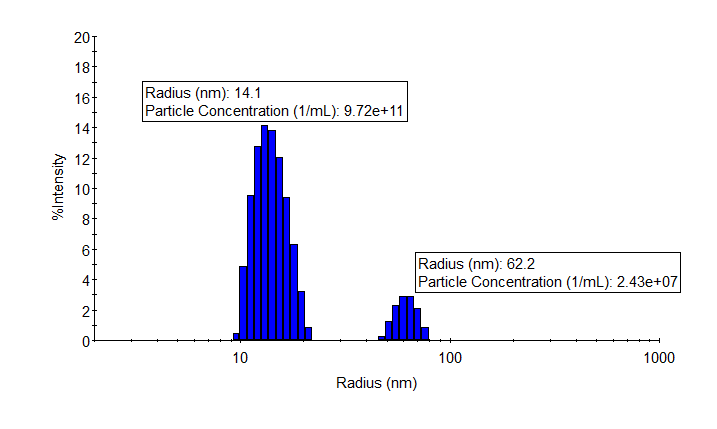
Partikel-Konzentration
Die Anzahl der Partikel pro mL ist eine kritische Information bei der Entwicklung von Nanopartikeln für therapeutische, diagnostische und weitere Anwendungen. DYNAMICS berechnet die Partikelkonzentration durch die Kombination von DLS und SLS: Die dynamische Lichtstreuung bestimmt die Partikelgröße, die kombiniert mit dem Brechungsindex des Materials und der gesamten Streuintensität ausreicht, um die Konzentration zu quantifizieren. Diese Messung kann auch bei Viren und Lipidnanopartikel angewendet werden.
Wie im Beispiel gezeigt, reichen wenige Mikroliter Probe und wenige Sekunden Messzeit aus, um Partikelgröße und Konzentration sowohl von Monomeren als auch von Aggregaten eines Adeno-assoziierten Virus zu bestimmen. Einfach können mehrere Serotypen getestet, Prozessparameter bewertet oder die Stabilität in Lagerpuffern beurteilt werden.
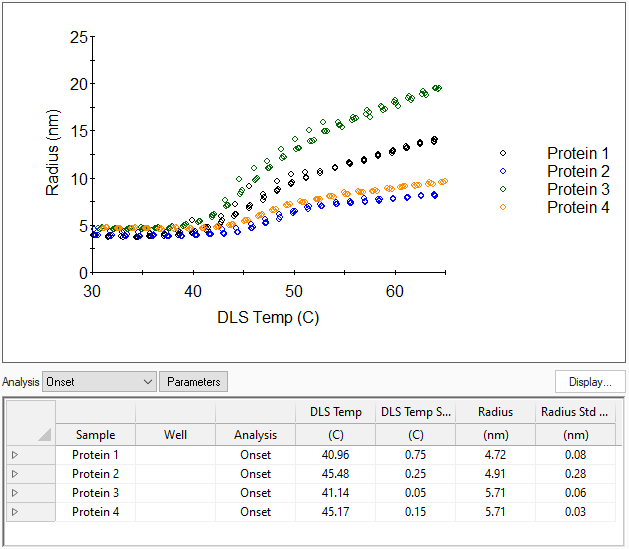
Thermische Stabilität - Schmelztemperatur, Aggregation, Entfaltung
Überwachen Sie das Schmelzen und Entfalten von Proteinen, bestimmen Sie die kritische Mizellentemperatur (CMT) oder untersuchen Sie temperatursensitive Polymere und Kolloide. DYNAMICS bestimmt die Übergangstemperaturen Tonset, Tagg und Tm, sowie die durchschnittliche Größe am Übergangspunkt.
Jedes Molekül verhält sich anders. Deshalb bietet DYNAMICS eine Analyse in Bezug auf die Onset-Übergangstemperatur für aggregierende Proben, sigmoide (Mittelpunkt) Konformationsänderungen wie z.B. die Entfaltung von Proteinen und eine Polynomanalyse für komplexere Vorgänge.
Kolloidale Stabilität - unspezifische Wechselwirkungen messen
Unspezifische Protein-Protein-Wechselwirkungen sind der Schlüssel für die Auswahl von biotherapeutischen Kandidaten wie IgG und die Optimierung ihrer Formulierung.
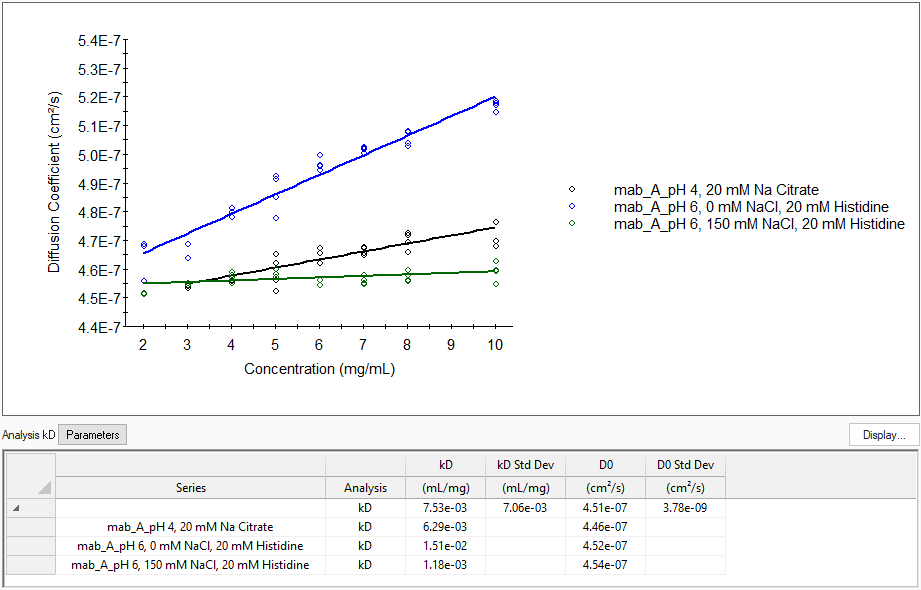
Durch Messung und Analyse einer Konzentrationsreihe können sowohl A2 (from static light scattering) und kD (aus dynamischer Lichtstreuung) bestimmt werden.
Dieses Beispiel zeigt die kD-Analyse einer Antikörperformulierung in verschiedenen Puffer- und Salzbedingungen. Die positiven Werte für kD weisen auf die Stabilität der Formulierung hin.
Es ist einfach, die kolloidale Stabilität eines monoklonalen Antikörpers unter verschiedenen Formulierungen zu bewerten, indem man eine Reihe von Konzentrationen und Bedingungen in einer Mikrotiterplatte testet. Dabei wird ein einziger Scan im DynaPro Plate Reader mit DYNAMICS durchführt. Zum Vergrößern anklicken.
Konzentrationsanalyse
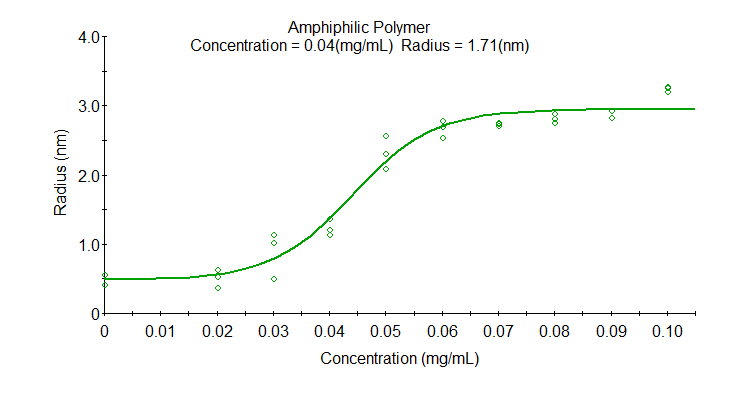
Bestimmen Sie die kritische Mizellenkonzentration (CMC) und andere konzentrationsabhängige Größenänderungen mit der Konzentrationsabhängigkeitsanalyse von DYNAMICS.
Das Beispiel zeigt die konzentrationsabhängige Größe für ein amphiphiles Polymer. Jede Konzentration wurde in Dreifachbestimmung im DynaPro Plate Reader gemessen. Die CMC, definiert als der Mittelpunkt des Übergangs, ist 0,04 mg/mL mit einem Radius von 1,7 nm.
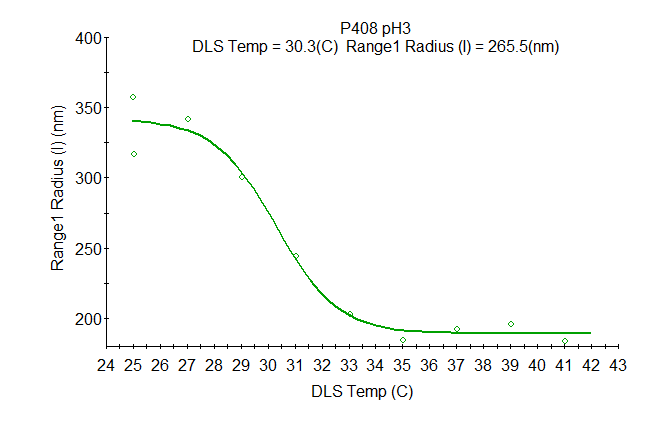
Parametrische Analyse für jeden Bedarf
Es gibt keine Einschränkungen, wenn es um die Analyse von Trends für Ihre Proben geht: Analysieren Sie jeden Übergang mit der benutzerdefinierten parametrischen Analyse. Wählen Sie Parameterpaare wie z. B. Radius gegen pH-Wert oder Zetapotential gegen Salzkonzentration. Temperatursensitive Polymere können wie in der Grafik gezeigt analysiert werden.
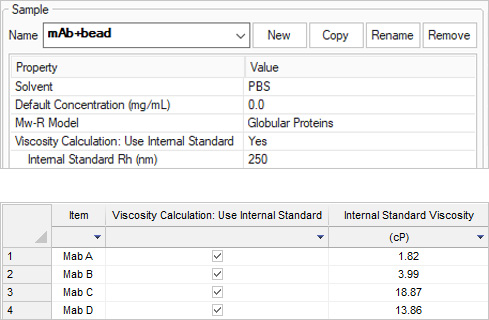
Viskositätsberechnungen
Die Lösungsviskosität von hochkonzentrierten Proteinformulierungen wird durch Messung der Diffusion eines Tracerpartikels bekannter Größe bestimmt. In DYNAMICS geben Sie einfach den hydrodynamischen Radius des Partikels mit Standardgröße an (250 nm in diesem Beispiel), setzen die Viskositätsberechnung auf „Use Internal Standard“ und die Viskosität der Formulierung wird berechnet und in der Datentabelle angezeigt.
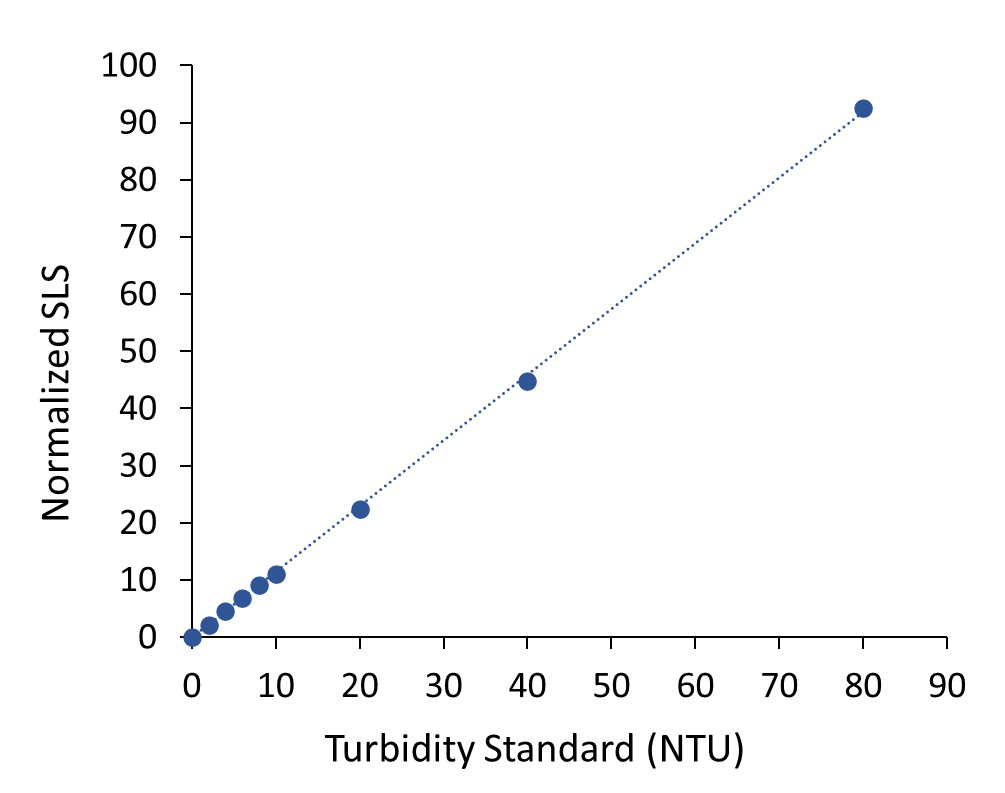
Trübung oder Opaleszenz
Die Opaleszenz einer Formulierung oder die Trübung einer Lösung wird durch Interpolation aus einer Trübungskalibrierkurve bestimmt. Anschließend wird die Probenlösung in dieser Küvette gemessen. Der gemessene Wert wird von Dynamics in nephekometrischen EIheiten angezeigt (NTU).
Besuchen Sie die Seite Trübung/Opaleszenz um mehr zu erfahren.