Konjugation & Beladungsgrad-Analyse
Die Analyse von makromolekularen Konjugaten in Lösung wird durch Lichtstreuung erheblich vereinfacht.
Einführung
Viele interessante und nützliche Makromoleküle bestehen aus zwei chemisch und physikalisch unterschiedlichen Spezies, die kovalent verbunden sind. Dies kann es recht schwierig machen, die wesentlichen Eigenschaften molarer Masse und Größe zu charakterisieren.
Die Kombination von DAWN® Mehrwinkel-Lichtstreu-Detektor (MALS) mit einem UV- und Optilab® Brechungsindex-Detektor (dRI) mit Größenausschlusschromatographie sowie der ASTRA Konjugatanalyse bietet die Lösung für die Charakterisierung konjugierter Makromoleküle. Dieser Aufbau kann zur Analyse des Konjugationsverhältnisses oder des Beladungsgrades sowie der Größenverteilungen, der Verkapselungseffizienz, der Form, der Konformation oder der Polymerverzweigung verwendet werden. Die Bestimmung der Parameter der einzelnen Komponenten und deren Abhängigkeit von der Größe kann für Arzneimittel, Impfstoffe und Gentherapien besonders wichtig sein.
Konjugierte Makromoleküle
Die herkömmliche analytische Größenausschlusschromatographie (SEC) reicht nicht aus, um Konjugate zu charakterisieren, da es keine Kalibrierstandards gibt, die das Elutionsverhalten des spezifischen Konjugats widerspiegeln. Die Kombination von MALS, UV- und dRI-Detektion mit SEC sowie dem ASTRA Algorithmus zur Konjugatanalyse bietet die Lösung für die Charakterisierung von Makromolekülen wie z. B.:
- Glykoproteine, Lipoproteine und Nanodiscs
- Protein-Polysaccharid-Impfstoffe
- Block-Copolymere
- PEGylierte Proteine
- Protein-Nukleinsäure-Komplexe
- In Detergenzmizellen solubilisierte Membranproteine
Funktionsprinzip
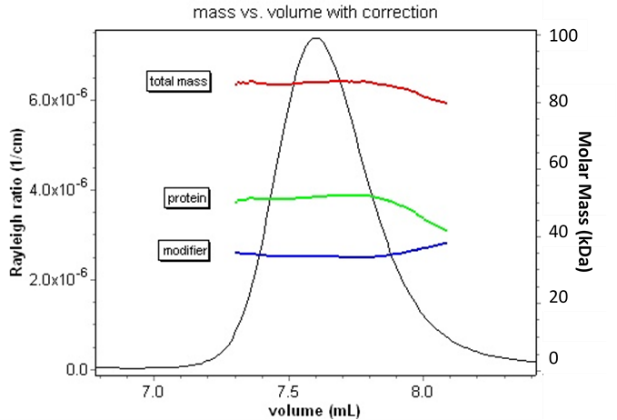
Konzeptionell kann ein Komplex, der aus einem Protein, das UV-aktiv ist und einem Modifikator wie Polyethylenglykol oder PEG, das keinen UV-Chromophor enthält, besteht, durch die Kombination von Informationen aus zwei verschiedenen Messungen analysiert werden. Die UV-Absorption bestimmt die Proteinkonzentration, während der RI die Gesamtkonzentration des Makromoleküls bestimmt. Diese beiden Messungen sind ausreichend, um das Verhältnis von Protein und Modifikator im Komplex zu bestimmen (dies trifft auch zu, wenn beide Komponenten UV-Licht absorbieren, solange die Extinktinktionskoeffizienten ausreichend verschieden sind).
Diese Analyse kann jedoch nicht die Gesamtmasse oder den oligomeren Zustand der Spezies bestimmen. Die Integration von MALS ermöglicht jedoch die vollständige Charakterisierung der Konjugate. Da die Lichtstreuung proportional zum Produkt aus molarer Masse und Konzentration ist, reicht die Kombination dieser drei Signale aus, um nicht nur das Verhältnis, sondern die tatsächliche molare Masse und damit den oligomeren Zustand jeder Komponente zu bestimmen. Diese Information ist entscheidend für die Beurteilung der Aggregation von Glykoproteinen, die Bestimmung der molekularen Konformation konjugierter Polysaccharide, die Messung der molaren Masse von Block-Copolymeren oder die Entscheidung, ob ein durch Detergenzien solubilisiertes Membranprotein als natives Oligomer vorliegt oder nicht.
AAV und kleine VLP
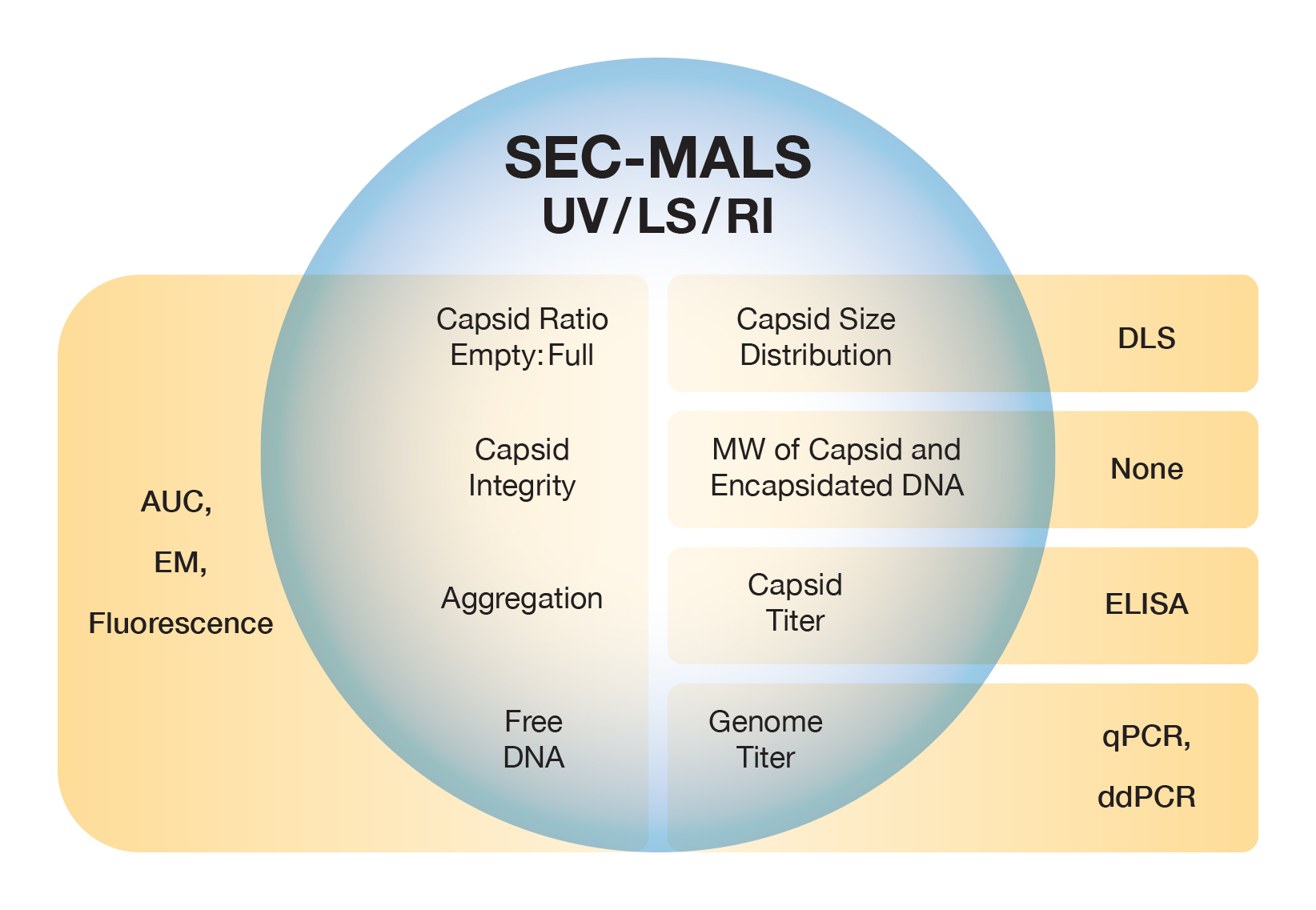
Adeno-assoziierte Viren und kleine, unbehüllte virusähnliche Partikel sind Sonderfälle der Standard-Konjugatanalyse. Ihre strukturellen Komponenten sind in der Regel sehr gut definiert in Bezug auf die Gesamtmolmasse der Proteine, die das Kapsid bilden und die genomische Beladung. Die SEC-MALS-UV-dRI-Methode bestimmt das Molekulargewicht der Kapside und das durchschnittliche Molekulargewicht der Beladung, woraus sich der Anteil der leeren und gefüllten Kapside berechnen lässt - auch wenn sie in der SEC zusammen eluieren. Bei Kenntnis der Extinktionskoeffizenten bei 260 nm und 280 nm, kann die gleiche Analyse in Kombination mit MALS durchgeführt werden.
Funktionsprinzip
Die ASTRA-Virusvektoranalyse erweitert die standardmäßige Konjugatanalyse, um nicht nur die molare Masse der Protein- und Nukleinsäurekomponenten zu bestimmen, sondern auch Vg/Cp, das Verhältnis der vollen Genome zu der gesamten Kapsidmenge. Des Weiteren kann die Gesamtkonzentration der leeren und vollen Kapside sowie deren Summe bestimmt werden. Außerdem wird der Aggregatanteil quantifiziert, so dass insgesamt drei kritische Qualitätsattribute (CQA) in einem einzigen Lauf quantifiziert werden.
Da die SEC-MALS-Methode unabhängig vom AAV-Serotyp ist, keine speziellen Reagenzien oder Prozeduren erfordert und mit Standard-HPLC-Instrumenten automatisiert werden kann, eignet sie sich als Plattformmethode für verschiedene AAV-Produkte und alle Phasen der Entwicklung, Herstellung und Qualitätskontrolle. Wyatt bietet eine umfassende Anleitung zur Entwicklung von Standardarbeitsanweisungen (SOP) für die AAV-Analyse mit SEC-MALS. Erfahren Sie mehr über die Charakterisierung von Genvektoren und die AAV-Servicepakete von Wyatt.
Nanokonjugate
Zusammengesetzte Nanopartikel bestehen im Allgemeinen aus einem Strukturmolekül und der Beladung. Wie bei konjugierten Makromolekülen ist die MALS-UV-dRI-Detektion mit einer Trenntechnik wie SEC oder Feldflussfraktionierung (FFF) eine wesentliche Methode zur Charakterisierung der Größe und Zusammensetzung solcher binären zusammengesetzten Nanopartikel. Aufgrund ihrer Größe ist die Analyse von Nanopartikeln mit diesem Verfahren jedoch mit Herausforderungen verbunden, die durch ASTRAs neuartigen Algorithmus zur Analyse von Nanokonjugaten überwunden wird. Zu den Produkten, die in diesen Größenbereich fallen - und für die Nanokonjugat-Analyse geeignet sind - gehören:
- Lipid-Nanopartikel (LNP), die RNA- oder DNA-Nutzlasten einkapseln
- Liposomen oder PLGA-Polyplexe, die kleine Wirkstoffe oder Peptide als Nutzlast enthalten
- Protein-Polysaccharid-Impfstoffe im Molmassenbereich von mehreren Megadalton
Funktionsprinzip
Die Konjugatanalyse beruht auf genauen UV-Messungen. Wenn der Radius des Partikels oder großen Makromoleküls über etwa 20 nm liegt, wird die Streuung des einfallenden UV-Lichts durch den Analyten selbst erheblich. Dies führt dazu, dass der Detektor ein UV-Extinktionssignal anzeigt, obwohl möglicherweise keine Absorption vorliegt. Ein leeres Liposom zum Beispiel absorbiert bei 260 nm kein UV-Licht, erzeugt aber aufgrund der Streuung ein nennenswertes UV-Signal. Eine unkorrigierte Verwendung dieser UV-Daten auf die Standard-Konjugatanalyse führt zu fehlerhaften Ergebnissen.
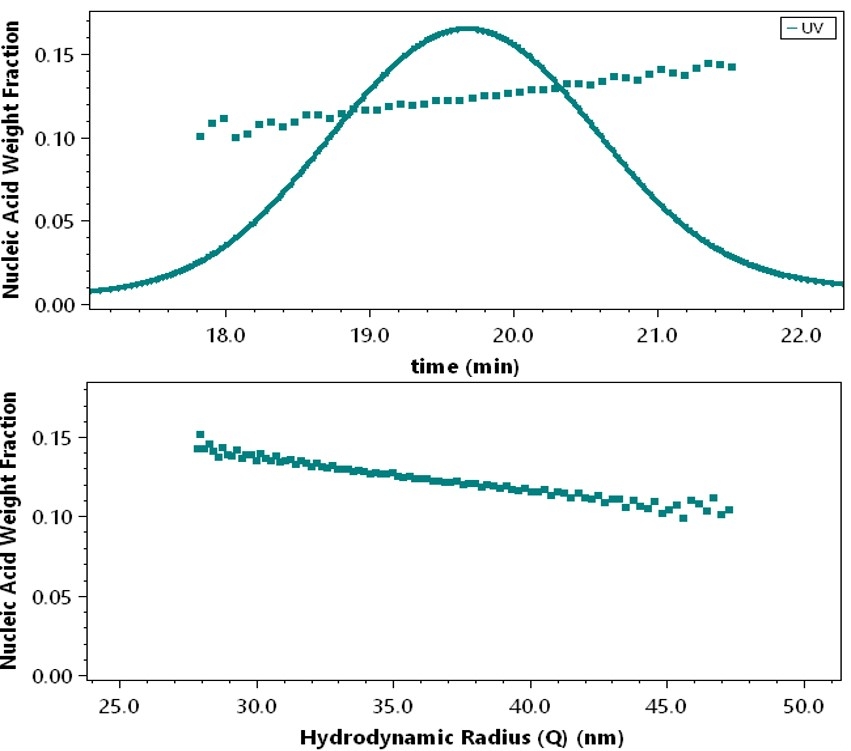
Die Nanokonjugat-Analyse charakterisiert das Signal des UV-Detektors durch die Streuung leerer Lipidpartikel oder nicht konjugierter Polysaccharide über einen Molmassenbereich, der die Größen der beladenen Nanopartikel oder konjugierten Polymere abdeckt. Diese Informationen werden dann auf einen firmeneigenen Algorithmus angewandt, um die Nukleinsäure- oder Wirkstoffbeladung oder das konjugierte Protein in der Probe zu quantifizieren. Da die Analyse bei jedem Elutionsvolumen erfolgt, wird die Beladung als Funktion der Größe bestimmt.
Instrumentierung
Die Nanokonjugat-Analyse kann in Verbindung mit der SEC verwendet werden, wenn die Proben in Bezug auf Größe und Stabilität mit dieser Methode kompatibel sind. Größere Partikel oder solche, die sich durch die Scherkräfte der Säule zersetzen könnten, können durch Feldflussfraktionierung (FFF) mit dem Eclipse™ FFF-System von Wyatt getrennt werden. Das Eclipse-System wird durch HPLC-Module nach Industriestandard unterstützt und lässt sich nahtlos mit den MALS- und dRI-Detektoren von Wyatt kombinieren.
SEC-MALS oder FFF-MALS mit ASTRAs LNP-RNA-Analyse ersetzen mehrere traditionelle Techniken für eine umfassende biophysikalische Analyse von LNPs, die Nukleinsäuren enkapsulieren, in einem einzigen Lauf.
Die LNP-RNA-Analyse liefert das Molekulargewicht der Nukleinsäure-Beladung bei jedem Elutionsvolumen und als Funktion der Größe.
Ein Eclipse FFF-MALS-System trennt und charakterisiert Makromoleküle und Partikel mit einer Größe von 1 bis 1000 nm.
Ressourcen
Webinare
Virtual User Meeting – Protein Conjugate and Copolymer Analysis
Quantify Viral Vector Attributes with Light Scattering
Analyzing Ebola virus glycoprotein and its interactions with therapeutic antibodies using CG-MALS
Characterizing Protein Conjugates and Their Aggregates by Light Scattering
Application Notes
Ausgewählte Literatur
Abbas, S.; Lodge, T. P. Depletion interactions: effects of added homopolymer on ordered phases formed by spherical block copolymer micelles. Macromolecules 2008, 41, 8895-8902.
Bensaid, F.; du Boullay, O. T.; Amgoune, A.; Pradel, C.; Reddy, L. H.; Didier, E.; Sablé, S.; Louit, G.; Bazile, D.; Bourissou, D. Y-Shaped mPEG-PLA cabazitaxel conjugates: Well-controlled synthesis by organocatalytic approach and self-assembly into interface drug-loaded core-corona nanoparticles. Biomacromolecules 2013, 14, 1189-1198.
Berguig, G. Y.; Convertine, A. J.; Shi, J.; Palanca-Wessels, M. C.; Duvall, C. L.; Pun, S. H., Press, O. W.; Stayton, P. S. Intracellular delivery and trafficking dynamics of a lymphoma-targeting antibody-polymer conjugate. Mol. Pharm. 2012, 9, 3506-3514.
Citkowicz, A. et al. Characterization of virus-like particle assembly for DNA delivery using asymmetrical-flow field-flow fractionation and light scattering. Analytical Biochemistry 376,(2) 163-172 (2008).
McIntosh, N.L. et al. Comprehensive characterization and quantification of adeno-associated vectors by size exclusion chromatography and multi angle light scattering. Scientific Reports 11: 3012, (2021)
Peng, Y.; Zhang, L. Characterization of a polysaccharide-protein complex from Ganoderma tsugae mycelium by size-exclusion chromatography combined with laser light scattering. J. Biochem. Bioph. Meth. 2003, 56, 243-252.
Slotboom, D. J.; Duurkens, R. H.; Olieman, K.; Erkens, G. B. Static light scattering to characterize membrane proteins in detergent solution. Methods 2008, 46, 73-82.
Werle, A.K. et al., Comparison of analytical techniques to quantitate the capsid content of adeno-associated viral vectors. Molecular Therapy: Methods & Clinical Development 23, (December 2021)
Instrumente für Konjugatanalyse
MALS Detektoren

DAWN® - Der empfindlichste MALS-Detektor, der auf dem Markt erhältlich ist. Beinhaltet Detektoren in 18 Winkeln zur Bestimmung der molaren Massen im Bereich von 200 Da bis 1 GDa und Radien von 10 - 500 nm.
- Standardoption: Umgebungstemperatur
- Option Heizen/Kühlen: -15 °C bis +150 °C
- Hochtemperatur-Option: Umgebungstemperatur bis +210 °C
Das DAWN bietet spezielle Optionen zur Messung fluorezierender Proben: Fluoreszenzsperrfilter und einen Infrarot-Laser mit 785 nm.
miniDAWN® - In der Empfindlichkeit nur vom DAWN übertroffen. Beinhaltet Detektoren in 3 Winkeln zur Bestimmung der molaren Massen im Bereich von 200 Da bis 10 MDa und Radien von 10 - 50 nm bzw. 150 nm, mit speziellen Streufunktionen. Nicht temperiert.
microDAWN® - Der erste MALS-Detektor für UHPLC, mit einer Interdetektor-Dispersion von nur 1,5 µL. Beinhaltet Detektoren in 3 Winkeln zur Bestimmung der molaren Massen im Bereich von 200 Da bis 20 MDa und Radien von 10 - 50 nm. Nicht temperiert.
Brechungsindex-Detektoren

Optilab® - Ein einzigartiges Online-Differenzialrefraktometer zur Konzentrationsmessung beliebiger Makromoleküle, unabhängig von Chromophoren. Die Hochkonzentrationsoption ermöglicht die Bestimmung von Proteinkonzentrationen bis zu 180 mg/mL.
microOptilab™ - Der erste RI-Detektor, der speziell für die Verwendung mit allen UHPLC-Systemen entwickelt wurde.
Feldflussfraktionierungssysteme

Die Feldflussfraktionierung führt vielseitige Trennungen von Makromolekülen und Nanopartikeln im Bereich von 1 bis 1000 nm im Radius und darüber hinaus durch. FFF-MALS-DLS ist das ideale Mittel, um Verteilungen von molarer Masse und Größe, Nanopartikelkonzentration und eine erweiterte Charakterisierung von Konformation und Konjugatgehalt zu erhalten.
Eclipse™ - FFF-Gerät mit überlegener Leistung, Zuverlässigkeit und Benutzerfreundlichkeit. Unterstützt das Dilution Control Module™ für erhöhte Empfindlichkeit und die SEC-Umschaltoption für die Nutzung des Systems im FFF- oder SEC-Trennungsmodus.
Mobility™ EAF4-System - Add-on zu einer Eclipse, das eine FFF mit elektrischen/asymmetrischen Fluss zur Bestimmung von Ladungs- und Zetapotentialverteilungen durchführt.
Eclipse-Kanäle - Die Eclipse bietet eine große Auswahl an Trennkanälen und Membranen, um eine Vielzahl von analytischen und sogar semipräparativen Aufgaben zu erfüllen.
Software
ASTRA® - Unsere umfassende Softwarelösung für die MALS-Analyse im Chromatographie-, FFF- oder Batch-Modus. ASTRA ist in einer 21CFR(11)-konformen Version erhältlich und bietet zusätzliche Optionen wie die Partikelanalyse.